
吴华君
邮箱:hjwu@bjmu.edu.cn
地址:北京市海淀区学院路38号
个人简介:
北京大学临床医学高等研究院PI,北京大学肿瘤医院研究员。本科毕业于北京师范大学生命科学学院,于中科院遗传与发育生物学研究所获得生物信息学博士学位,曾在美国哈佛大学Dana-Farber癌症研究院先后任职博士后,助理研究员,数据科学家。
实验室主页:http://wulab.bjmu.edu.cn
主要研究方向:
我们开发用于多组学数据分析和整合的生物统计和机器学习算法,并指导其在人类发育,衰老和疾病研究中的应用。我们的研究主要围绕以下几个方面展开:
(1)3D基因组建模研究基因表观调控和非编码变异功能
3D基因组通过基因表观调控参与重要的生物学过程,其异常改变与发育和多种重大疾病相关。染色质构象捕获(Hi-C)技术是解析3D基因组结构的关键技术,Hi-C技术产生的高通量数据由于数据量大结构复杂,在分析上存在很大挑战。我们设计了基于线性模型的Hi-C数据校正算法,并开发了Hi-C数据的整合分析软件HiCapp(Bioinformatics,2016)。而后,我们建立了基于Hi-C数据鉴定全基因组CTCF环结构域的计算方法,并将两个软件结合分析了多种组织和细胞的Hi-C数据,发现单基因CTCF环结构域内部包含的拓扑隔离基因富集于组织发育和细胞分化等重要的生物学过程,提出拓扑隔离基因可能的进化过程(图 1),并利用模型证明了单基因CTCF环结构域和其内部的基因在干细胞发育过程中具有重要的生物学功能(Nature Communications,2021)。
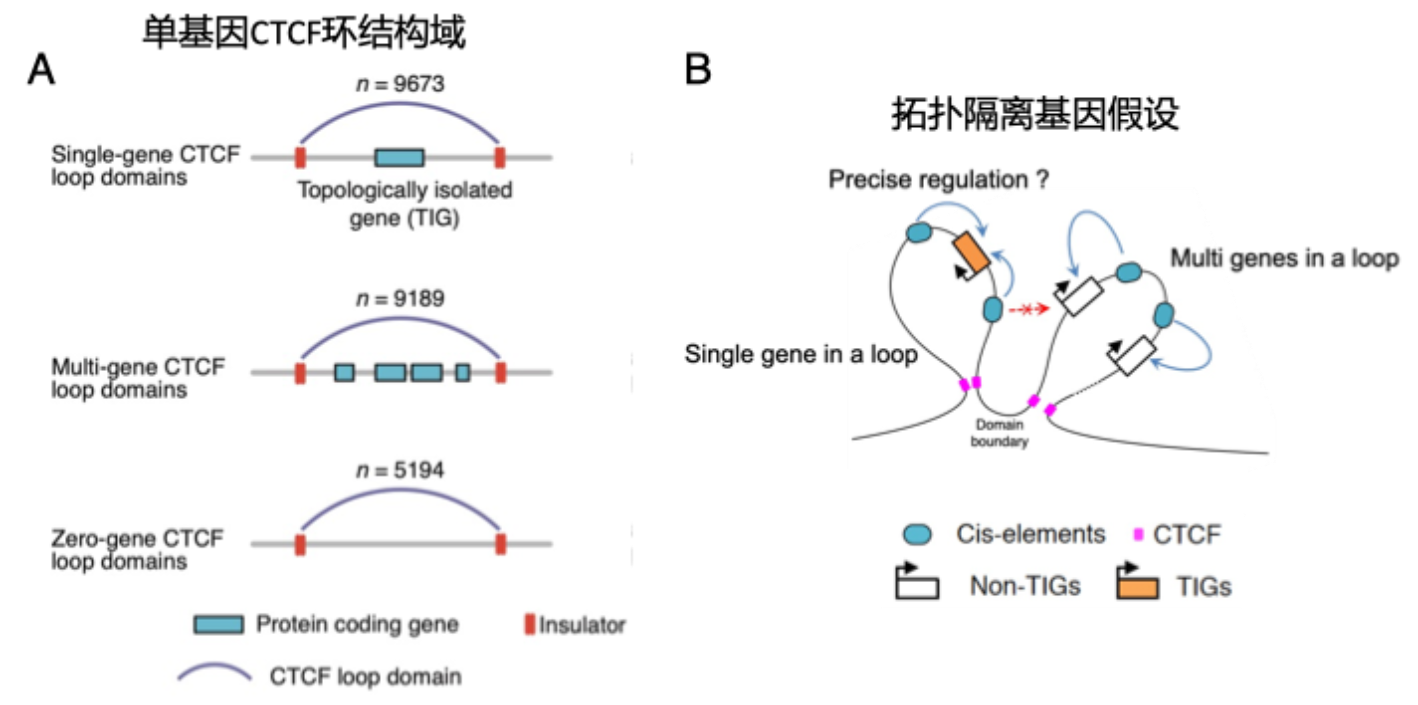
图 1. 单基因CTCF环结构域和拓扑隔离基因。
(2)单细胞多组学和空间组学数据分析的机器学习算法及应用
近年来,随着单细胞技术的成熟,超千万细胞级别的单细胞数据集的出现,对目前的单细胞聚类算法提出了挑战。我们开发了一种基于二部图的单细胞数据高效聚类算法Secuer,实现单个CPU在3分钟内完成对超大型数据集(1千万个细胞)的无监督聚类任务,在保持准确度的同时,运行速度比目前的通用算法快10倍,使用的内存资源比通用算法少10倍(图 2)。以Secuer为基本单元的基于二部图的集成聚类算法Secuer-consensus在损失一些计算效率的同时可以提高聚类的鲁棒性,适用于对单细胞数据的精准聚类分析(PLOS Computational Biology,2022)。
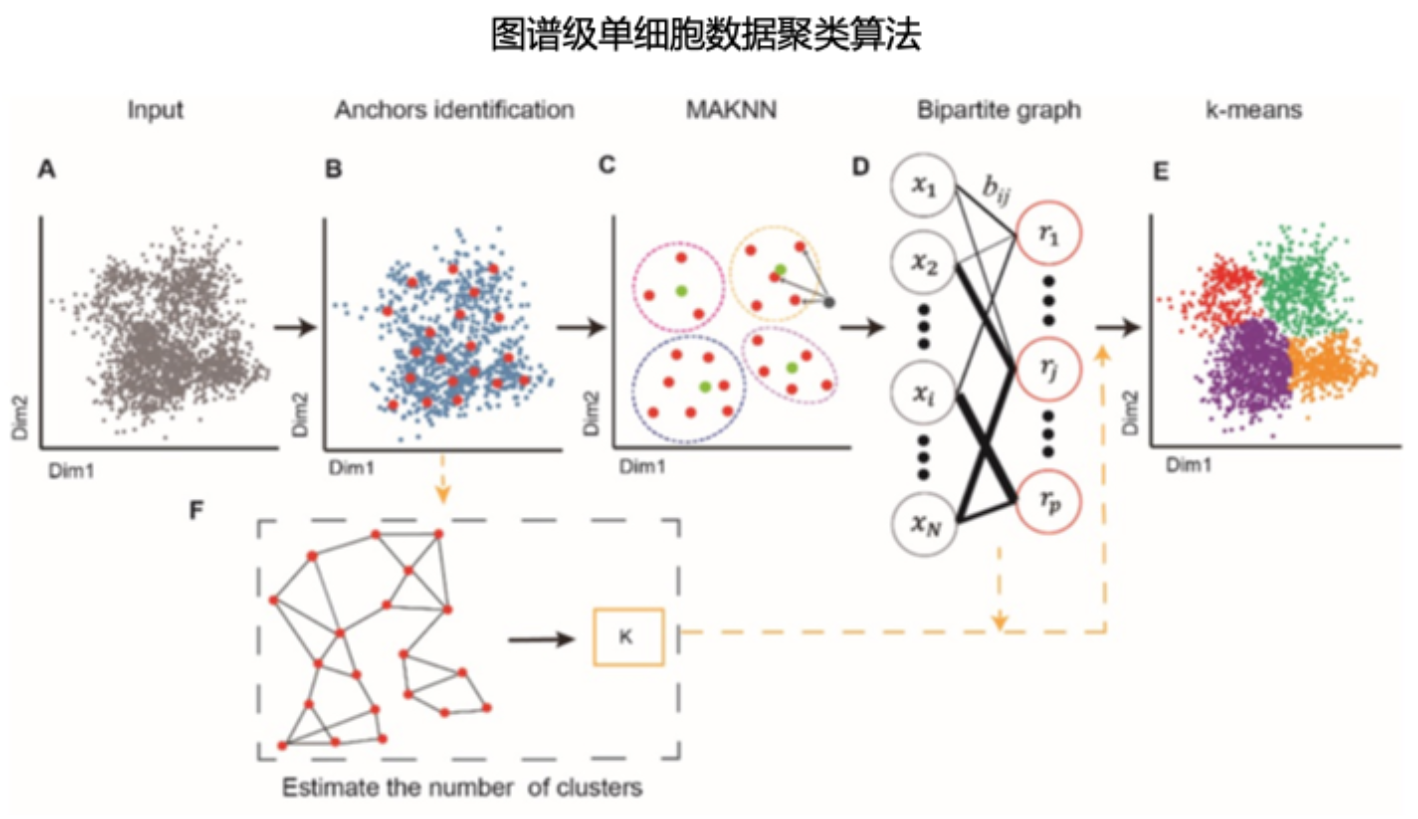
图 2. 基于二部图的单细胞数据聚类算法Secuer。
(3)开发多组学算法预测多种生物过程中的关键调控因子
我们基于多种肿瘤空间组学数据,提出了肿瘤内空间异质性的计算方法和分类标准,定义了肿瘤内细胞的两种空间异质性模式:聚集模式(Clustered)和随机模式(Random),并发现随机模式的癌症患者具有更高的复发几率和死亡风险(图 3)。多组学数据分析发现随机模式的肿瘤具有细胞粘附力不足、免疫激活和特有的肿瘤微环境等特征,并探讨了这两种空间异质性模式可能是由DNA拷贝数变异的克隆型决定。本研究中我们利用多种空间组学技术,结合新的计算方法,构建肺腺癌中肿瘤内异质性的空间图谱,对肿瘤内空间异质性的机制和与临床预后的关系提出了新的见解,为空间多组学技术在癌症中的研究提供了新的思路(Cell Genomics,2022)。
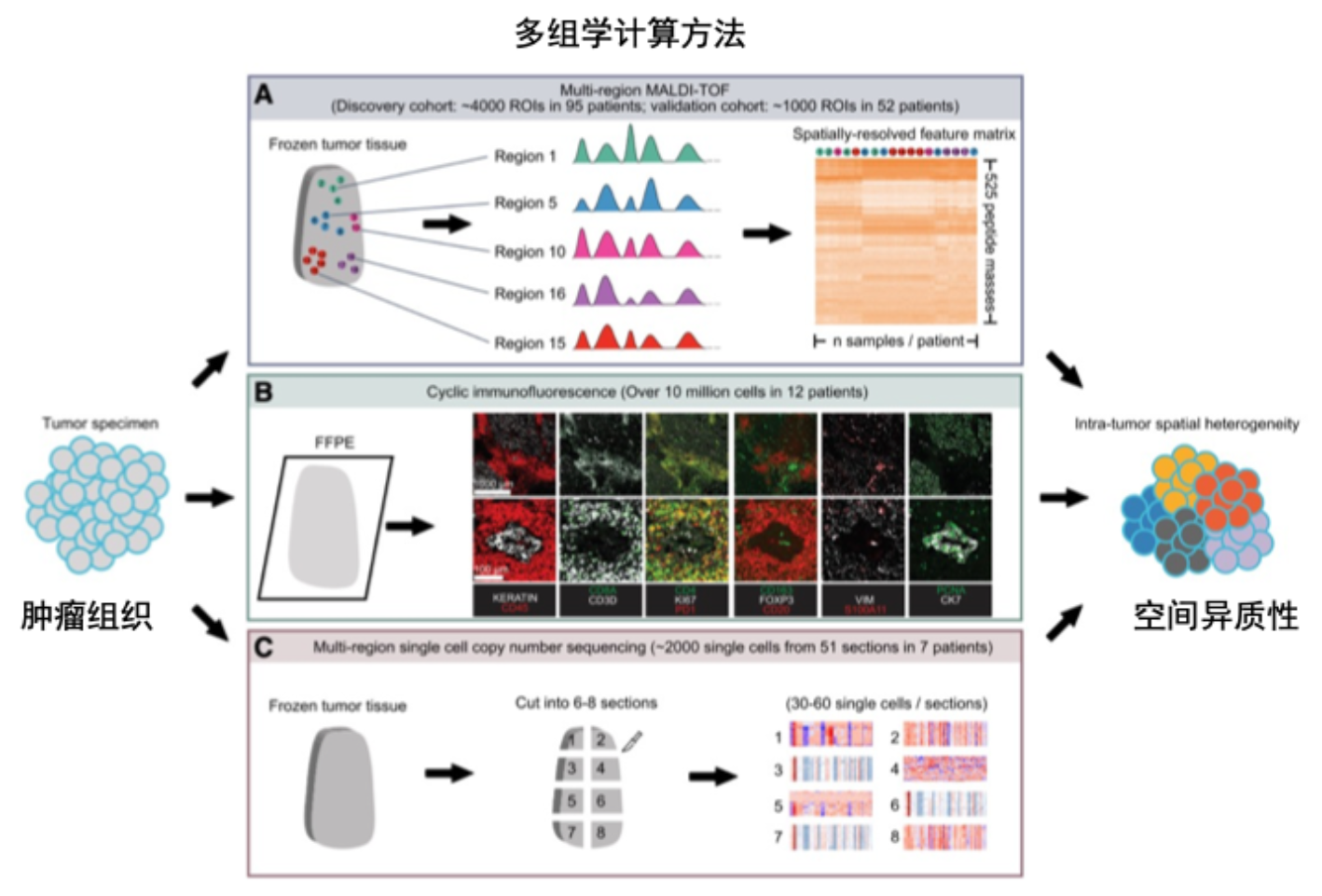
图 3. 空间多组学技术解析肿瘤内空间异质性。
代表性科研项目:
国家自然科学基金面上项目(2023 - 2026,2025 - 2028)
北京市自然科学基金面上项目(2024 - 2026)
科技部国家重点研发计划(2021 - 2023)
北京大学临床医学+X青年专项(2022)
10篇代表性论文:
(1) Zhong C*, Jiang WJ*, Yao Y, Li Z, Li Y, Wang S, Wang X, Zhu W, Wu S, Wang J, Fan S, Ma S, Liu Y, Zhang H, Zhao W, Zhao L, Feng Y, Li Z, Guo R, Yu L, Pei F, Hu J, Feng X, Yang Z, Yang Z, Yang X, Hou Y, Zhang D, Xu D, Sheng R, Li Y, Liu L, Wu HJ#, Huang J#, and Fei T#, CRISPR screens reveal convergent targeting strategies against evolutionarily distinct chemoresistance in cancer. Nature Communications, 2024. 15(1): p. 5502.
(2) Wang G*, Wang H*, Ji X*, Wang T, Zhang Y, Jiang W, Meng L, Wu HJ#, Xing X#, and Ji J#, Intratumoral microbiome is associated with gastric cancer prognosis and therapy efficacy. Gut Microbes, 2024. 16(1): p. 2369336.
(3) Jiang WJ, Hu C, Lai F, Pang W, Yi X, Xu Q, Wang H, Zhou J, Zhu H, Zhong C, Kuang Z, Fan R, Shen J, Zhou X, Wang YJ, Wong CCL#, Zheng X#, and Wu HJ#, Assessing base-resolution DNA mechanics on the genome scale. Nucleic Acids Research, 2023. 51(18): p. 9552-9566.
(4) Wei N, Nie Y, Liu L#, Zheng X#, and Wu HJ#, Secuer: Ultrafast, scalable and accurate clustering of single-cell RNA-seq data. PLOS Computational Biology, 2022. 18(12): p. e1010753.
(5) Wu HJ*, Temko D*, Maliga Z*, Moreira AL, Sei E, Minussi DC, Dean J, Lee C, Xu Q, Hochart G, Jacobson CA, Yapp C, Schapiro D, Sorger PK, Seeley EH, Navin N, Downey RJ#, and Michor F#, Spatial intra-tumor heterogeneity is associated with survival of lung adenocarcinoma patients. Cell Genomes, 2022. 2(8).
(6) Wu HJ*, Landshammer A*, Stamenova EK, Bolondi A, Kretzmer H, Meissner A#, and Michor F#, Topological isolation of developmental regulators in mammalian genomes. Nature Communications, 2021. 12(1): p. 4897.
(7) Wu HJ and Michor F, A computational strategy to adjust for copy number in tumor Hi-C data. Bioinformatics, 2016. 32(24): p. 3695-3701.
(8) Shu SK*, Wu HJ*, Ge JY, Zeid R, Harris IS, Jovanovic B, Murphy K, Wang BB, Qiu XT, Endress JE, Reyes J, Lim K, Font-Tello A, Syamala S, Xiao TF, Chilamakuri CSR, Papachristou EK, D'Santos C, Anand J, Hinohara K, Li W, McDonald TO, Luoma A, Modiste RJ, Nguyen QD, Michel B, Cejas P, Kadoch C, Jaffe JD, Wucherpfennig KW, Qi J, Liu XS, Long H, Brown M, Carroll JS, Brugge JS, Bradner J, Michor F#, and Polyak K#, Synthetic Lethal and Resistance Interactions with BET Bromodomain Inhibitors in Triple-Negative Breast Cancer. Molecular Cell, 2020. 78(6): p. 1096-+.
(9) Jun HJ*, Appleman VA*, Wu HJ*, Rose CM, Pineda JJ, Yeo AT, Delcuze B, Lee C, Gyuris A, Zhu HH, Woolfenden S, Bronisz A, Nakano I, Chiocca EA, Bronson RT, Ligon KL, Sarkaria JN, Gygi SP, Michor F, Mitchison TJ#, and Charest A#, A PDGFR alpha-driven mouse model of glioblastoma reveals a stathmin1-mediated mechanism of sensitivity to vinblastine. Nature Communications, 2018. 9.
(10) Hinohara K*, Wu HJ*, Vigneau S, McDonald TO, Igarashi KJ, Yamamoto KN, Madsen T, Fassl A, Egri SB, Papanastasiou M, Ding L, Peluffo G, Cohen O, Kales SC, Lal-Nag M, Rai G, Maloney DJ, Jadhav A, Simeonov A, Wagle N, Brown M, Meissner A, Sicinski P, Jaffe JD, Jeselsohn R, Gimelbrant AA, Michor F#, and Polyak K#, KDM5 Histone Demethylase Activity Links Cellular Transcriptomic Heterogeneity to Therapeutic Resistance. Cancer Cell, 2018. 34(6): p. 939-953 e9.
