
跨学部生物医学工程系
赖彬彬
邮箱:laib@bjmu.edu.cn
地址:北京市海淀区学院路38号
个人简介
赖彬彬博士,2008年获中国吉林大学数学与应用学士学位;2013年获北京大学生物力学与医学工程博士学位; 2014至2019年于美国国立卫生研究院NIH从事博士后研究工作。2020年1月份起加入北京大学医学部跨学部生物医学工程系(现临床医学高等研究院)任研究员、博士生导师,入选国家海外高层次人才引进计划(青年项目),北医博雅青年学者,同时兼任北京大学第一医院研究员,北大国际癌症研究院PI,分子肿瘤学国家重点实验室PI。
主要研究方向
1、单细胞表观基因组学方法研发
研发过包括单细胞微小核酸酶测序(scMNase-seq)、Trac-looping三维染色质开放结构测序等方法,现在正在研发单细胞表观组学多模态测序方法。例如,开发的单细胞微小核酸酶测序方法首次绘制了单细胞分辨率小的核小体展位图谱,并揭示了全新的核小体占位规律和增强子预备阶段的染色质开放的胞间异质性,成果发表于Nature上。

图1:新方法揭示不同染色质状态下的核小体占位规律
2、利用单细胞组学技术研究正常与疾病状态下的皮肤细胞异质性和发病机制
利用RNA-seq,scRNA-seq,ATAC-seq,scATAC-seq,scMultiome,CUT&Tag等组学方法绘制包括人体全身不同部位正常皮肤细胞以及多种皮肤疾病状态下(包括Keloid,BCC,melanoma,和EMPD等)皮肤细胞的转录组和表观组,揭示不同部位皮肤细胞的差异的分子基础,皮肤疾病和皮肤肿瘤的发生发展机制。已有成果发表于JID等期刊上。
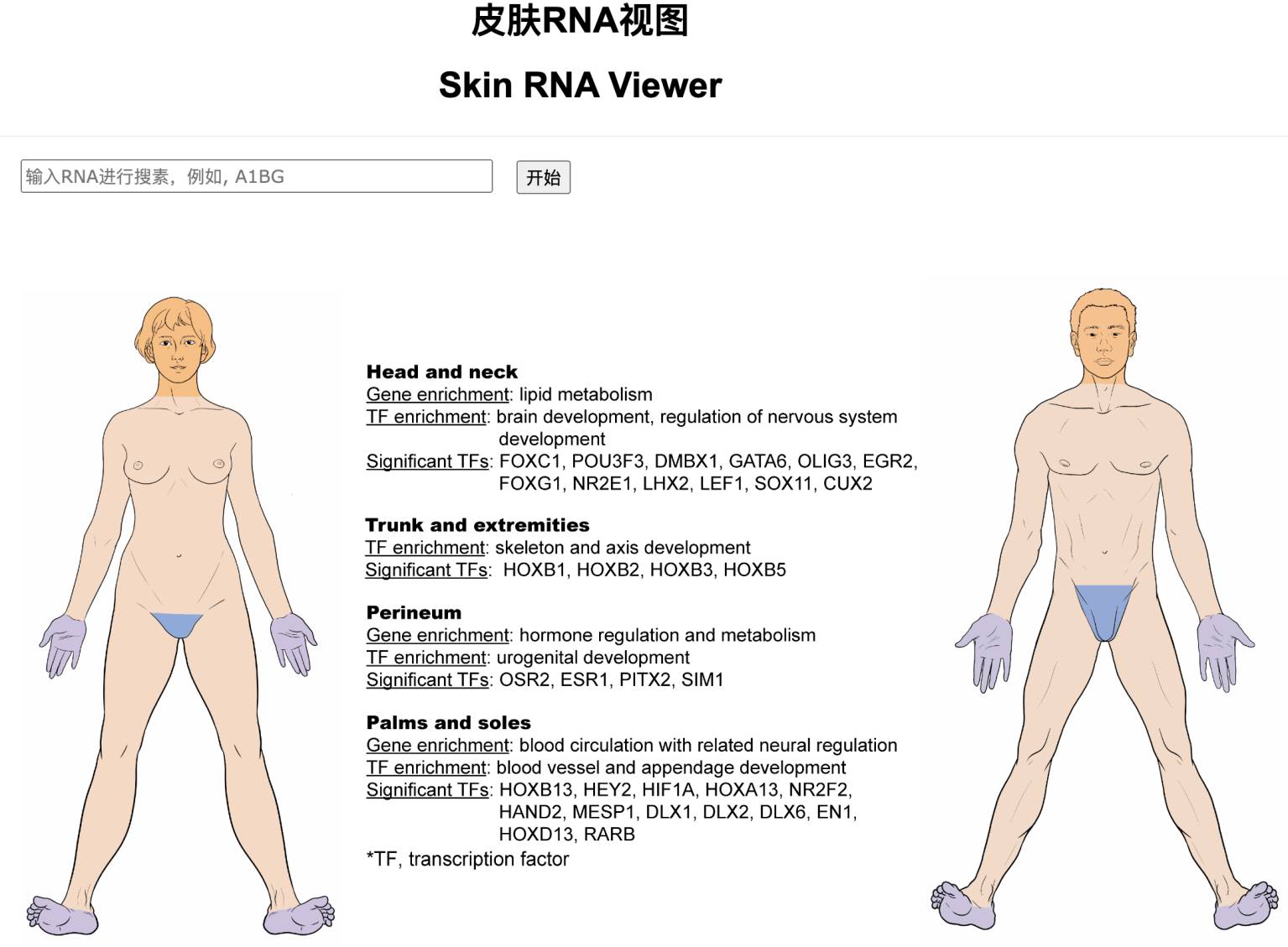
图2. 正常皮肤不同部位的分子特征
3、组学大数据分析方法的研发和应用
在诱导干细胞的生物材料研究中,针对如何预测诱导之后的干细胞是否分化成了特定的细胞类型的生物学问题,我们开发了结合机器学习和RNA-seq的干细胞身份预测方法MED-P。主要是通过对已有的数据库中的分化细胞的转录组数据进行学习,再把学到的模型对新的数据进行预测。已有结果发表于Advanced Materials上。
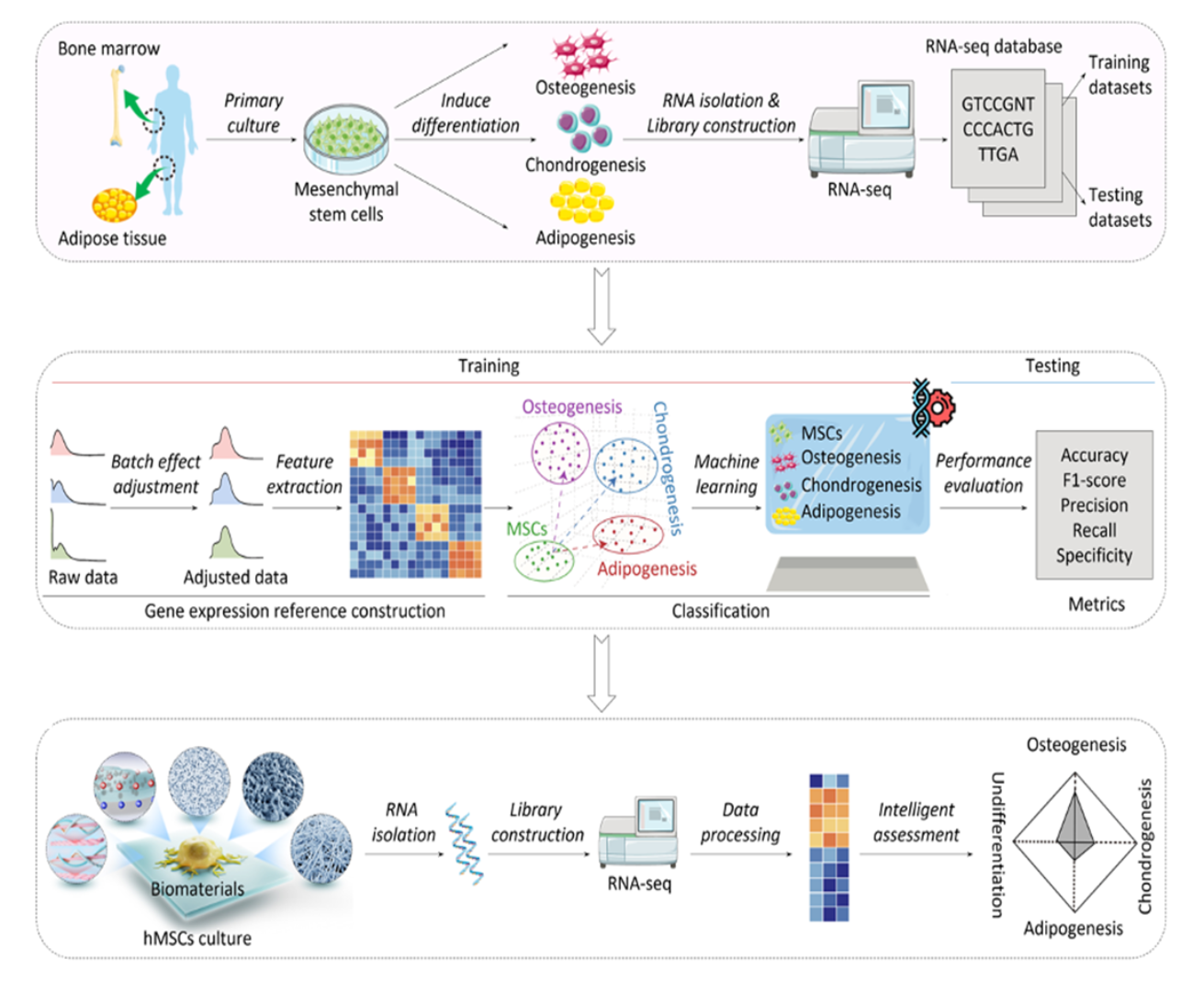
图3. 利用大数据智能预测干细胞分化功能的研究流程图。
代表性科研项目
2021-2024 NSFC面上项目,负责人,“单细胞核小体占位图谱的生物信息学方法研究”
2023-2025 科技部国家重点研发计划,负责人,“单细胞表观基因组多模态测序技术研发与在皮肤衰老和肿瘤 中的应用”
2023年度 北京大学“临床医学+x”青年专项,负责人,“利用高通量及单细胞测序研究口腔纳米材料细胞周期毒性的分子机制”
10篇代表性论文
Zhou, Y*., Ping, X.*, Guo, Y.*, Heng, B. C., Wang, Y., Meng, Y., Jiang, S., Wei, Y., Lai, B.#, Zhang, X. #, & Deng, X#. Assessing Biomaterial-induced Stem Cell Lineage Fate by Machine Learning-based Artificial Intelligence. Advanced Materials, 35:2210637. (2023).
Yan, Y., Tian, J., Wang, Y., Li, Y., Zhang, C., Zhang, S., Lin, P., Peng, R., Zhao, C., Zhuang, L., Lai, B. #, Zhou, L. #, Zhang, G. #, & Li, H. # Transcriptomic heterogeneity of skin across different anatomic sites. Journal of Investigative Dermatology, 143(13):398-407.e5 (2022).
Ren G*, Lai B*, Harly C, Baek S, Ding Y, Zheng M, Cao Y, Cui K, Yang Y, Zhu J, Hager GL, Bhandoola A, Zhao K. Transcription factors TCF-1 and GATA3 are key factors for the epigenetic priming of early innate lymphoid progenitors toward distinct cell fates. Immunity, 55(8):1402–1413.e4. (2022).
Hu L, Zhao X, Li P, Zeng Y, Zhang Y, Shen Y, Wang Y, Sun X, Lai B#, Zhong C#. Proximal and Distal Regions of Pathogenic Th17 Related Chromatin Loci Are Sequentially Accessible During Pathogenicity of Th17. Front Immunol. 13:864314 (2022).
Gao W, Lai B, Ni B#, Zhao K#. Genome-wide profiling of nucleosome position and chromatin accessibility in single cells using scMNase-seq. Nat. Protocol, 15:68-85 (2020).
Harly C, Kenney D, Ren G, Lai B, Raabe T, Yang Q, Cam MC, Xue HH, Zhao K, Bhandoola A. The transcription factor TCF-1 enforces commitment to the innate lymphoid cell lineage. Nat Immunol, 20(9):1150-1160 (2019).
Lee JE, Schmidt H, Lai B, Ge K. Transcriptional and Epigenomic Regulation of Adipogenesis. Mol Cell Biol, DOI: 10.1128/MCB.00601-18 (2019).
Lai B, Gao W, Cui K, Xie W, Tang Q, Jin W, Hu G, Ni B, Zhao K. Principles of nucleosome organization revealed by single-cell micrococcal nuclease sequencing. Nature, 562:281-285 (2018).
Lai B*, Tang Q*, Jin W*, Hu G*, Wangsa D, Cui K, Stanton BZ, Ren G, Ding Y, Zhao M, Liu S, Song J, Ried T, Zhao K. Trac-looping measures genome structure and chromatin accessibility. Nat Methods, 15(9):741-747 (2018).
Lai B*, Lee JE*, Jang Y, Wang L, Peng W#, Ge K#. (2017) MLL3/MLL4 are required for CBP/p300 binding on enhancers and super-enhancer formation in brown adipogenesis. Nucleic Acids Res, 45(11):6388-6403.
